Une nouvelle compréhension du fonctionnement des enzymes pourrait orienter l’avenir de la production de médicaments
Des chercheurs de la Faculté de médecine de l’Université McGill ont fait des progrès importants vers l’élucidation du fonctionnement d’enzymes qui jouent un rôle essentiel dans la production d’antibiotiques et d’autres médicaments. Leurs résultats sont publiés dans la revue Science.
« Une bonne partie des médicaments sur lesquels nous comptons à l’heure actuelle sont d’origine naturelle, produits par la flore de notre planète », explique le professeur agrégé Martin Schmeing, du Département de biochimie de McGill, auteur principal de l’étude. « Il s’agit entre autres de molécules fabriquées à l’intérieur de microbes par des enzymes massives appelées synthétases peptidiques non ribosomiques, ou NRPS. Les NRPS synthétisent toutes sortes d’antibiotiques qui peuvent tuer des bactéries ou des champignons dangereux, ou nous aider à combattre des infections virales et des cancers. Il s’agit par exemple de la viomycine, un antibiotique utilisé pour traiter la tuberculose multirésistante, de la cyclosporine, employée comme immunosuppresseur pour les greffes d’organes, ou encore d’un antibiotique familier comme la pénicilline. »
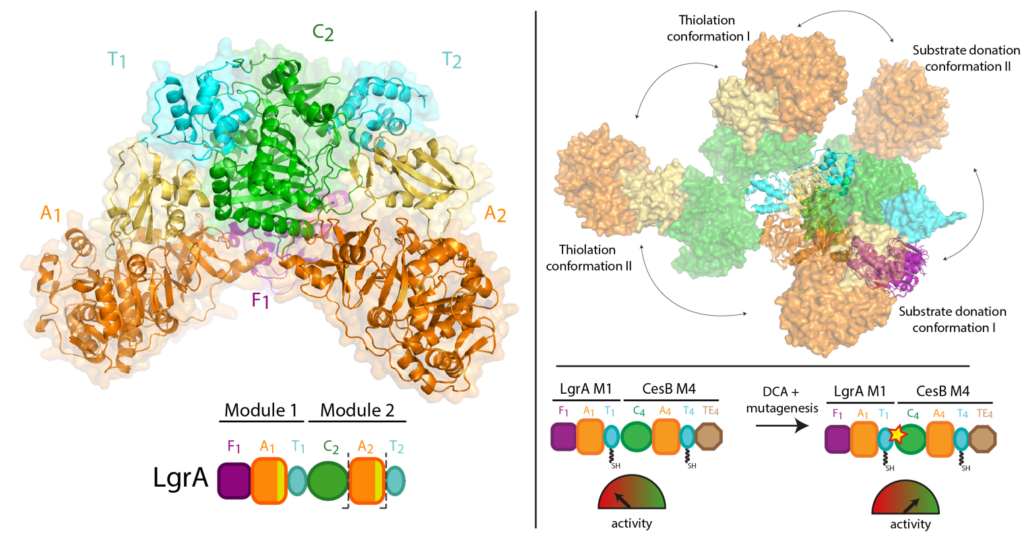
Pour synthétiser ces médicaments, les NRPS fonctionnent comme des chaînes de montage composées d’une série de machines. Chaque machine, appelée « module », exécute un procédé en plusieurs étapes au moyen de parties mobiles qui lui permettent d’ajouter un élément à la molécule en construction.
Percer les mystères de la chaîne de montage
Les travaux antérieurs du Pr Schmeing et d’autres chercheurs ont permis d’élucider le fonctionnement d’un de ces modules. Aujourd’hui, au moyen d’une technique appelée cristallographie aux rayons X utilisée au Centre canadien de rayonnement synchrotron, en Saskatchewan, ainsi qu’au centre Advanced Photon Source en Illinois, l’équipe a pu prendre des photos 3D à ultra haute résolution des NRPS.
Pour la première fois, les chercheurs ont pu observer précisément l’interaction entre un module individuel et le reste de la chaîne de montage, en visualisant une portion de deux modules des NRPS qui fabriquent la gramicidine linéaire (un antibiotique qu’on trouve dans les produits Polysporin). L’équipe a constaté un manque étonnant de synchronisation entre les modules, sauf au moment où ils doivent se coordonner pour faire passer le peptide intermédiaire d’un module à l’autre. Les chercheurs ont aussi découvert que les modules ne se plaçaient ni en ligne droite ni d’aucune façon organisée, s’alignant plutôt dans de nombreuses positions relatives différentes. « Nous ne nous attendions pas à un tel degré de flexibilité », ajoute le Pr Schmeing, également directeur du Centre de biologie structurale de l’Université McGill. « Les enzymes font toute une gymnastique. »
Puisqu’elle observait les protéines immobilisées dans un cristal, l’équipe a pris soin de confirmer que ses résultats étaient représentatifs de la réalité. Le Pr Schmeing a travaillé avec sa collègue, la professeure Alba Guarné, du Département de biochimie, pour valider ces observations au moyen de données complémentaires sur les protéines en solution recueillies au centre Advanced Light Source de Berkeley. « La communauté de recherche en biologie structurale à McGill est très solidaire. Nous nous entraidons dans le cadre de collaborations, pour obtenir l’équipement biophysique nécessaire à nos expériences de pointe, et pour former nos étudiants », note le Pr Schmeing, soulignant le talent de Janice Reimer, Max Eivaskhani et Ingrid Harb, les doctorantes et doctorant mcgillois qui ont réalisé les expériences de l’étude. « L’environnement et les collègues du Centre de biologie structurale de McGill sont importants pour assurer le succès continu de nos laboratoires. »
Les résultats pourraient avoir une incidence, à terme, sur la production de nouveaux antibiotiques et médicaments. Depuis la découverte des NRPS, les scientifiques rêvent de créer des NRPS par bio-ingénierie, en combinant différents modules de façon à produire des molécules sur mesure. « Notre étude révèle qu’il devrait être possible de combiner ces modules comme on le souhaite, mais il y a un bémol : pour que les NRPS conçues par bio-ingénierie fonctionnent correctement, il faut les modifier aux points qui sont impliqués dans le passage du peptide en construction d’un module à un autre », conclut le Pr Schmeing. « C’est ce que nous avons fait en validation de principe dans le cadre de cette étude, en collaboration avec Martin Weigt de la Sorbonne, mais le processus devra être optimisé pour pouvoir produire des médicaments sur mesure. »
L’article « Structures of a dimodular nonribosomal peptide synthetase reveal conformational flexibility », par Reimer, J. M., Eivaskhani, M, Harb, I., Guarne, A, Weigt, M., Schmeing, T. M. est paru dans la revue Science le 8 novembre 2019. Doi:10.1126/science.aaw4388
Le 8 novembre 2019
